南科大吴柘、戴紫薇团队合作揭示植物RNA Pol II在基因3’端停顿的调控机制及意义
日期:2023-09-26
近日,南方科技大学生命科学学院副教授吴柘课题组和助理教授戴紫薇课题组合作在国际知名学术期刊Genome Biology发表了题为“Coupling of co-transcriptional splicing and 3’ end Pol II pausing during termination in Arabidopsis”的研究论文,通过实验结合数学建模,系统探究了模式植物拟南芥RNA Pol II转录终止阶段在基因3’端发生停顿的特征和调控规律,揭示了Pol II在基因3’端的停顿是确保植物多内含子基因高效共转录剪接的重要节点。
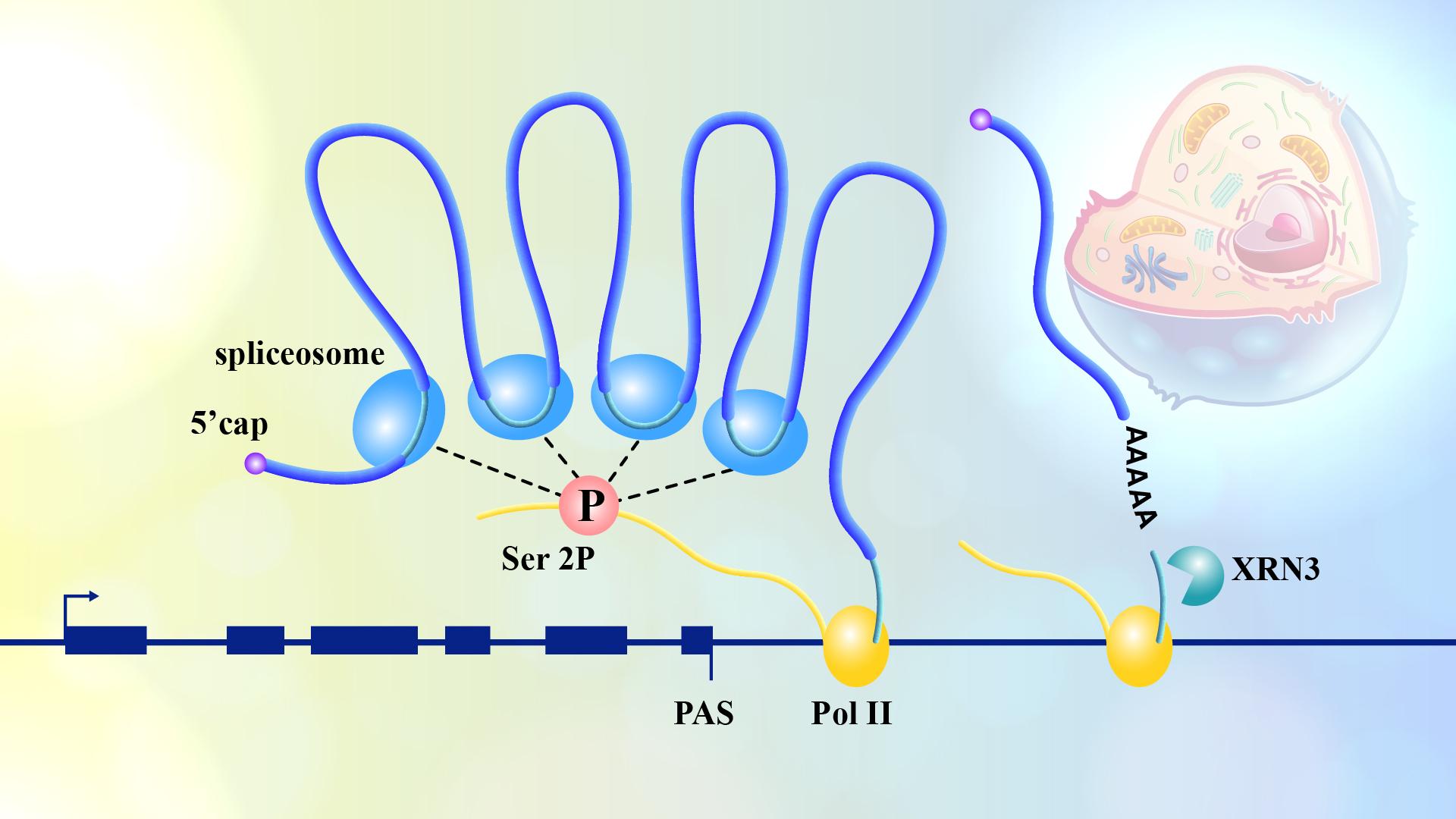
转录及其偶联的RNA加工过程是真核生物遗传信息解码中的核心事件,是基因型决定表型的关键。在植物中,转录因子通过调节转录起始从而调控各种生物学过程的机制已经为人熟知。然而,对转录延伸和终止阶段的调控不论是机制还是生物学意义在植物中还不甚清楚。此外,动植物中RNA加工均与转录紧密偶联,典型的例子是转录同时发生的RNA剪接,也称作共转录剪接(co-transcriptional splicing)。共转录剪接不仅会移除内含子,也会同时递送一些蛋白标记剪接过的RNA, 从而进一步调控RNA在转录后的命运(如降解和出核等)。植物基因通常内含子较短,平均长度仅为哺乳动物的1/10,然而植物的pre-mRNA仍然可以发生高效的共转录剪接,而其背后的机制不清楚1。此外,区别于哺乳动物,植物的Pol II在临近polyA位点下游很窄的区间内会发生转录停顿(3’端停顿)2,此前普遍认为这种停顿反映了植物基因相对高效的转录终止,但3’停顿与转录终止过程究竟有何机制上的联系以及如何被调控都不清楚。
研究首先利用单碱基分辨率的pNET-seq(plant Native Elongating Transcripts Sequencing)技术2,获得了Pol II在基因3’端分布的高精度图谱,发现Pol II在基因3’端停顿是拟南芥基因转录的一个重要特征,并且用3’停顿系数(3’ Pause Index, 3’ PI)定义了Pol II在基因3’端停顿的强度。经分析发现Pol II在基因3’端停顿的位置而非强度与转录终止效率相关,而且转录终止缺陷的突变体xrn3中Pol II在基因3’端的停顿并没有受到强烈影响,表明Pol II在基因3’端的停顿虽然与转录终止密切偶联,但是仍有其他因素调控Pol II在基因3’端停顿的强度。
进一步分析发现Pol II在基因3’端的停顿程度和基因上包含的外显子个数呈现强相关性(图1a, b, c),外显子数目越多的基因,Pol II在3’端停顿的程度越高。有趣的是,此前吴柘课题组报道了基因上包含的外显子数目也紧密关联到共转录剪接的效率,外显子越多的基因倾向于具有越高的共转录剪接效率1。因此,本研究对Pol II在基因3’端停顿强度、外显子个数以及共剪接效率三者间进行了进一步的相关性分析,发现三者之间存在相互依赖的正相关性(图1d),暗示了基因的外显子个数以及共转录剪接效率可能是影响Pol II在基因3’端停顿的重要因素。
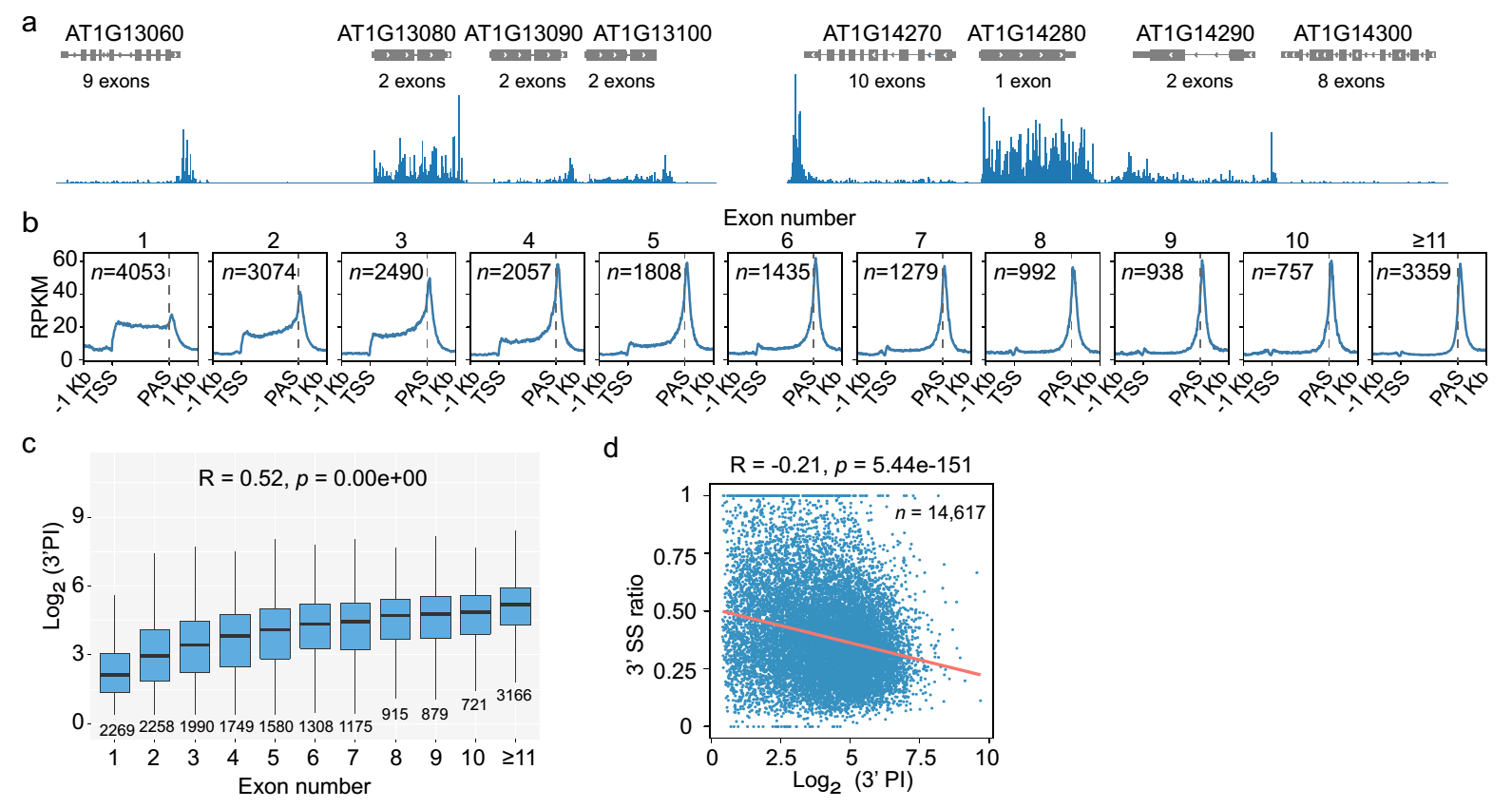
图1:Pol II在基因3’端停顿的强度和基因的外显子个数及共转录剪接效率呈正相关。
进一步,研究利用herboxidiene处理植物,抑制剪接复合体的组装进而抑制剪接,发现抑制剂处理后Pol II在基因3’端停顿的水平剧烈降低,并且基因外显子数目和Pol II 3’端停顿强度间的相关性不复存在(图2),显示剪接体的有效组装对Pol II 在基因3’端停顿是必要的。
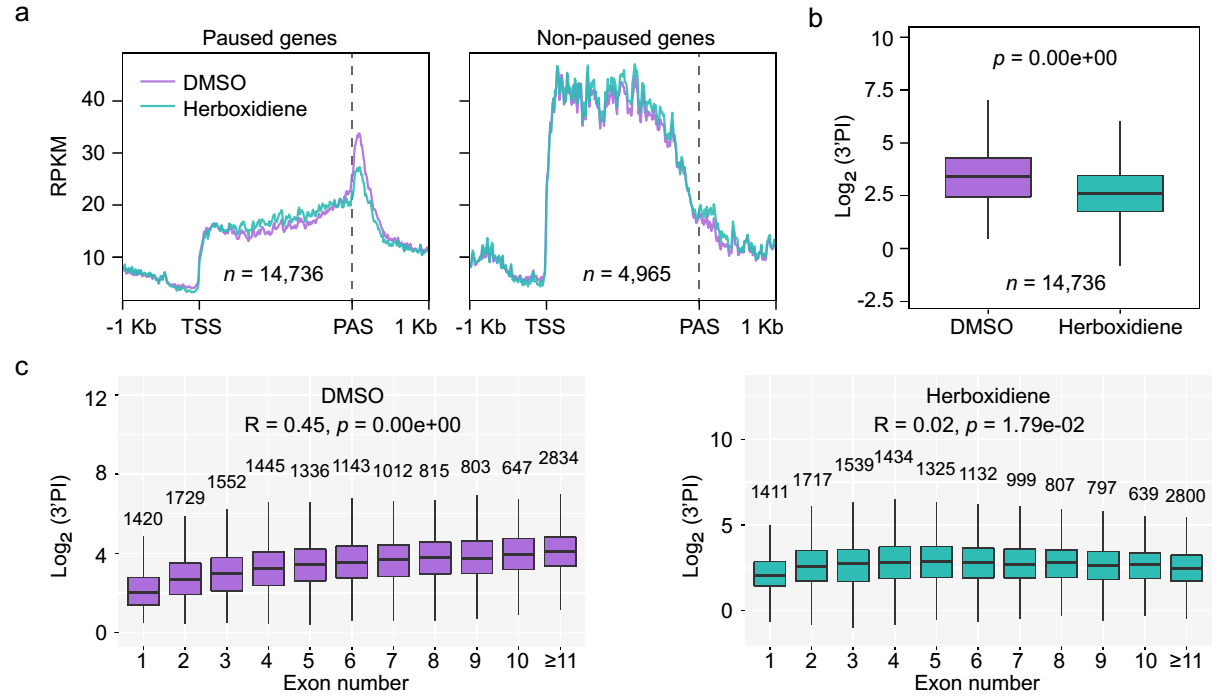
图2:剪接抑制剂herboxidiene处理降低了Pol II在基因3’端停顿的水平,并且破坏了3’端的停顿水平和外显子个数的相关性。
最后,研究构建了模拟Pol II转录延伸及终止过程的数学模型,并利用模型拟合了实验中获得的Pol II分布数据,通过拟合的结果确定和比较了不同条件及不同基因型中Pol II转录过程中的几种动力学参数。拟合结果进一步支持了外显子个数是影响Pol II在基因3’端停顿时间长短的重要因素这一结论。
作为结论,研究提出基因的外显子数目和共转录剪接偶联并决定了Pol II在基因3’端停顿的强度,剪接复合体的有效组装是Pol II在基因3’端停顿的必要条件,而Pol II在基因3’端的停顿可能为多外显子基因高效的共转录剪接提供保障(图3)。研究明确了植物Pol II在基因3’端停顿的意义和调控规律,为植物转录终止调控机制提供了新的线索。
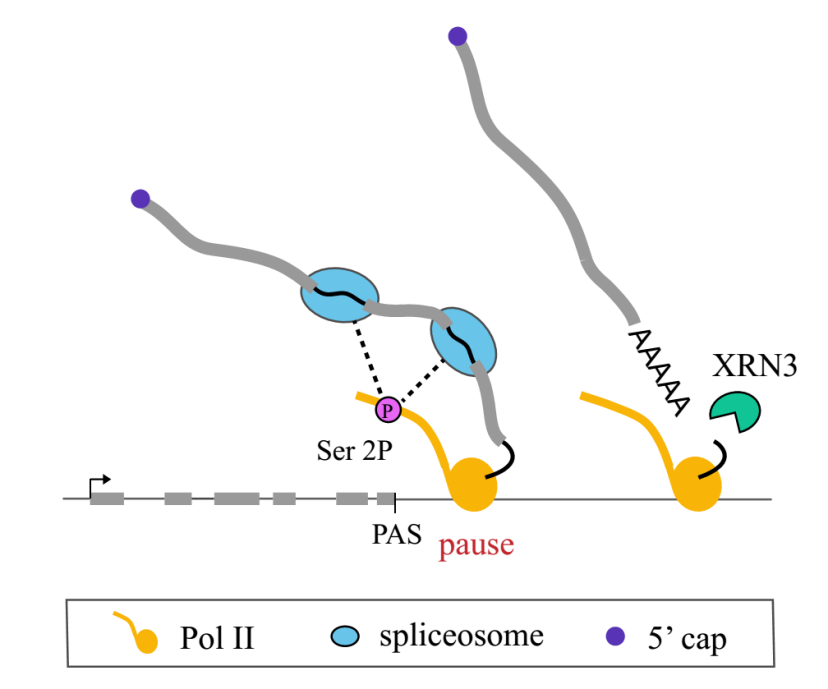
图3:拟南芥Pol II在基因3’端停顿的调控机制模型。
吴柘课题组博士生周思娴和博士后赵凤利(现中国水稻所助理研究员)为该论文的共同第一作者。吴柘和戴紫薇课题组分别完成了论文的实验和数学建模部分,为论文的共同通讯作者,南科大研究副教授朱丹灵和博士生张琦琦也参与了本项研究。该研究得到了国家自然科学基金、广东省创新创业团队项目、深圳市科创委以及广东省细胞工厂分子设计重点实验室基金的资助。
论文链接:https://doi.org/10.1186/s13059-023-03050-4
参考文献:
1. Zhu D, Mao F, Tian Y, Lin X, Gu L, Gu H, Qu LJ, Wu Y, Wu Z: The Features and Regulation of Co-transcriptional Splicing in Arabidopsis. Mol Plant 2020, 13:278-294.
2. Zhu J, Liu M, Liu X, Dong Z: RNA polymerase II activity revealed by GRO-seq and pNET-seq in Arabidopsis. Nat Plants 2018, 4:1112-1123.
供稿:生命科学学院
通讯员:付文卿
主图:丘妍
编辑:朱增光
最新动态
-
南科大 Andrew P. Hutchins 团队揭示 BRD8 调控干细胞多能性状态的核心机制
Date:2024-12-25
-
生命科学学院召开2024年度年会
Date:2024-12-17
-
南科大宋毅团队揭示植物塑造抗旱微生物组的遗传和生态学机制
Date:2024-12-10
-
南科大陈小菁课题组揭示海马空间认知功能的发育机制
Date:2024-12-04
